26 标准主轴分析介绍
本部分主要来自于对 Warton 等 (2012) 和 Warton 等 (2006) 内容的理解。我姑妄言之,各位也就姑妄听之,从一个取巧的角度来讲,我觉得可以从下面的角度理解标准主轴分析:
我们要比较差异,如果没有分组,我们要比较我们数据的斜率或截距是否等于一个值,例如,要研究异速生长关系,那么斜率肯定不能等于 1 吧,不然 y 和 x 是相等的生长速度,哪里来的异速生长?虽然我的文章我应该有 9 年不看了,我记忆里,是否为异速生长,就是拿斜率和 1 来比较,看其是否差异显著,当然,显著差异是我需要的。
我们要分析几个分组的数据,单纯的采用线性回归是没法比较的,他们肯定是斜率和解决不同,相同那不就是同一组数据了,这样我们就要检验通过一个主轴的斜率和截距来比较,如果存在共同的斜率,那我们只能在比较截距,如果截距存在差异,某一个要高,就接着分析这个差异可能的因素就行了,这个让我毕业的文章里是用的什么,我就记不清楚了,反正就分析了什么海拔生境之类的得出的结论,我也懒得再看了,反正我用不到了。
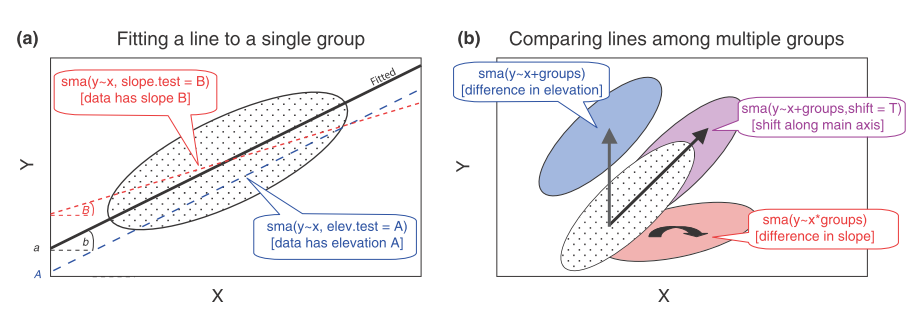
根据我多年前的记忆,SMA 的用途可以用下图进行表达:
-
单个分组的检验,分为斜率和截距的两种:
- 单个分组的斜率:为检验斜率是否等于某个值 b,一个简单的方法来检验就是当使用 b 作为斜率时,检验残差和轴的得分是否无关。
- 拟合的直线经过样品的中心点 (\bar x, \bar y),计算的截距接近正态分布,使用 t 检验来验证真正的截距是否等于某个特定值 a。
-
多个分组(多条直线)检验,是否存在共同斜率和共同截距
共同斜率:共同斜率检验是检验解决是否有偏移的前提,也就是说,根据我的理解,结果存在共同斜率,我们写文章是要找差异的,找不到差异了,我们只能退而求其次,来找其截距的不同。当然,没有共同斜率,比截距也没意义。
共同截距:如果存在共同斜率,我们接着要比较其是否存在共同截距。
如果上面两个都相同怎么办?那还有一个检验可以来做,那就是其会沿着主轴的方向发生偏移, Warton 等 (2006) 解释了这种情况,两个物种的叶片性状在主轴方向上偏移,原因是不同营养状况下他们的叶片寿命的差异。
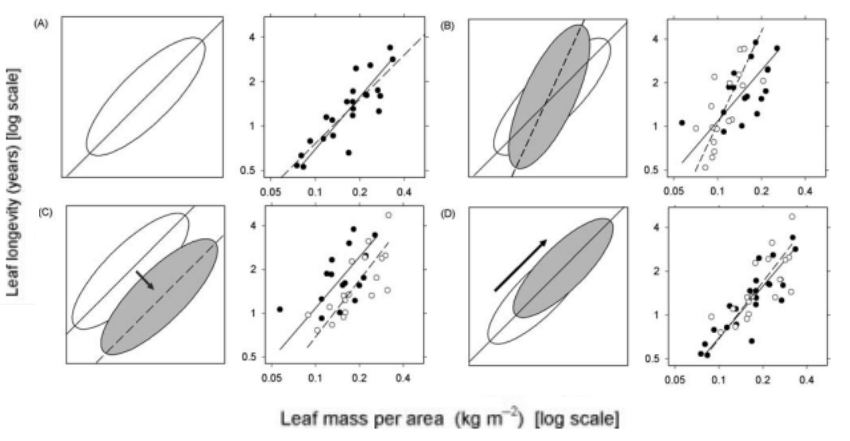
虽然 Warton 等 (2012) (Figure 26.1) 的图是彩图,但这个 Warton 等 (2006) 分开的图形更直观的解释了上面的几种情况:
- A: 检验斜率是否等于特定值
- B:检验斜率是否不同
- C:检验截距是否不同
- D:检验是否沿着主轴方向有偏移
26.1 sma 函数的参数介绍
我还是使用软件包自带的数据,然后结合 Warton 等 (2012) 文章的内容,在尽量详细解释一下参数的意思和返回的结果,期望能使得各位不会的能够看懂。
这个软件包的最重要的函数为 sma,
其参数的意义下面就分开详细解释
26.1.1 formula
R 中的 formula 对象,通过不同的写法可以得到不同的目的,具体如下面的叙述。
单样本检验
这里但从名字上即可以看出,这应该属于上图 A 的检验,这里使用的 formula 的形式为:
sma(y~x),拟合 SMA 并构造真正斜率和截距的的置信区间。sma(y~x, slope.test = B),检验 SMA 的斜率是否为 B。sma(y~x, elev.test = A),检验 SMA 的截距是否为 Asma(y~x, robust = T),使用 Huber 的 M 估计进行拟合,我对这种回归不了解,不过应该属于稳健回归的范畴,也就是帮助文档里所讲的,对野点的处理比较好。sma(y~x-1), 强制从原点通过的拟合。也就是这时候没有截距的差异,或者对截距的差异不感兴趣。
多个样本检验
也就是对多个直线进行比较,有多种分组的方式:
sma(y~x*groups),也就是检验所有的分组有没有共同斜率。sma(y~x+groups, type = "elevation"),检验具有共同斜率的分组有无共同截距。sma(y~x*groups, type = "shift"),检验斜率是否沿着主轴方向有漂移。sma(y~x+groups, slope.test = "B"),检验共同斜率是否为 B。sma(y~x+groups, elev.test = "A"),建议共同截距是否为 A。sma(y~x*groups-1),同单个样本的检验,也可以检验主轴方向的漂移。
多重比较
当 multcomp = TRUE 时,会进行各个水平间的多重比较,包括斜率、截距和沿着主轴的漂移(取决于使用的 formula)。
拟合结果
讲到这里应该继续讲函数的参数,不过我觉有必要在这里趁热打铁了,因为我觉得讲完了 formula 的几种形式,SMA 就讲完了,我们有必要把拟合结果顺带解释一下:
coef:拟合的参数,如果有多个样品的比较,将返回模型的参数,例如检验共同斜率时各个样品或者分组的斜率
nullcoef:零假设的参数。
alpha:见下文的参数解释
method:拟合使用的方法, ’MA’ 或 ’SMA’。
intercept:拟合的线是否强制通过原点。
call:调用 ma 或 sma 函数。
data: 见下文的参数
log: 见下文的参数
variables: SMA 的线使用的变量
origvariables:如有在拟合前有转换,转换前的变量列表
groups:分组变量的水平
gt:grouptest 缩写,表示分组测试的类型
gtr:分组测试的结果
slopetest:斜率假设检验的结果,以列表形式输出,包括 p 值、 检验统计、共同斜率 b 和置信区间 ci。
elevtest:截距假设检验的结果,返回 p 值、t检验统计、共同截距和它的置信区间。
slopetestdone:是否进行斜率检验
evevtestdone:是否进行斜率的检验
n:样品的大小
r2:决定系数
pval:统计结果的 p 值
from, to:在作图
plot.sma作图时使用的拟合线的的端点groupsummary:以数据框整理的分组参数
无需详细解释的参数
sma 中其他无需详细解释的参数:
data: 包含 x 和 y 变量的数据框
subset:用于拟合的数据框的子集,非必须选项。
na.action: R 中对 na 值处理的方式,例如删除等。
log:对变量使用以 10 为底的对数转换。
method:如果等于 SMA 为标准主轴分析,MA 为主轴分析,或者选择 OLS,最小二乘法。
type:如果多条线进行比较,我们应该选择的选项,如 elevation 或 shift。
alpha:我们置信区间里使用的 \alpha,默认 0.05。
slope.test: 假设检验使用的值,也就是与某一个值差异是否显著时使用。
elev.test:与 slope.test 类似,只不过是检验截距。
robust:如果为 TRUE,使用文件回归分析,目前仅针对单样本检验。
V:测量误差的变异矩阵,默认无。
n_min:一个分组最小的样品数量
quiet:如果为 TRUE,不提示任何信息
multcomp:如果为 TRUE,则进行多重比较
multcompmethod: 多重比较 p 值的方法。
26.2 自带数据的例子
安装从 CRAN 上进行即可:
Code
install.packages('smatr')我们来使用自带的数据进行分析示例,先加载需要的软件包和数据:
对于我们生态学人来说,数据浅显易懂,地点,降雨量,土壤 P 含量,叶片寿命、比叶面积几个数据:
Code
kable(head(leaflife), caption = "示例数据{#tbl-sma-data}")| site | rain | soilp | longev | lma |
|---|---|---|---|---|
| 1 | high | high | 1.1145511 | 125.48736 |
| 1 | high | high | 0.5161786 | 82.28108 |
| 1 | high | high | 0.9718517 | 71.02316 |
| 1 | high | high | 0.6722023 | 94.66730 |
| 1 | high | high | 1.0947123 | 119.70161 |
| 1 | high | high | 2.0606299 | 205.82589 |
26.2.1 单个样品的分析
我感觉单个样品的分析用的比较少,但不排除有些情况适用,我们看一下下面的结果
Code
Call: sma(formula = longev ~ lma, data = leaflife, log = "xy")
Fit using Standardized Major Axis
These variables were log-transformed before fitting: xy
Confidence intervals (CI) are at 95%
------------------------------------------------------------
Coefficients:
elevation slope
estimate -2.698800 1.315031
lower limit -3.224305 1.096261
upper limit -2.173295 1.577457
H0 : variables uncorrelated
R-squared : 0.4544809
P-value : 4.0171e-10 Code
Call: sma(formula = ..1, data = ..4, log = "xy", method = "MA", slope.test = 1)
Fit using Major Axis
These variables were log-transformed before fitting: xy
Confidence intervals (CI) are at 95%
------------------------------------------------------------
Coefficients:
elevation slope
estimate -3.085214 1.492616
lower limit -3.968020 1.146777
upper limit -2.202407 2.001084
H0 : variables uncorrelated
R-squared : 0.4544809
P-value : 4.0171e-10
------------------------------------------------------------
H0 : slope not different from 1
Test statistic : r= 0.3515 with 65 degrees of freedom under H0
P-value : 0.0035393 上面的结果 H0 的 p 值仅为 0.003,也就是发生的概率太小,我们拒绝 H0,有显著差异,实际上斜率为 1.49,也很明显。
26.2.2 多个样品的分析
我们先对降雨量低的数据的性状进行分析:
Code
Call: sma(formula = longev ~ lma * soilp, data = low_rain, log = "xy")
Fit using Standardized Major Axis
These variables were log-transformed before fitting: xy
Confidence intervals (CI) are at 95%
------------------------------------------------------------
Results of comparing lines among groups.
H0 : slopes are equal.
Likelihood ratio statistic : 3.943 with 1 degrees of freedom
P-value : 0.047076
------------------------------------------------------------
Coefficients by group in variable "soilp"
Group: high
elevation slope
estimate -2.523634 1.1825381
lower limit -3.135266 0.9398479
upper limit -1.912003 1.4878965
H0 : variables uncorrelated.
R-squared : 0.7392636
P-value : 1.462e-07
Group: low
elevation slope
estimate -3.837710 1.786551
lower limit -5.291926 1.257257
upper limit -2.383495 2.538672
H0 : variables uncorrelated.
R-squared : 0.80651
P-value : 0.00041709 我们看到 H0 : slopes are equal 的概率是 0.047,也就是无共同斜率,图形也佐证了该点:
Code
plot(diff_rain)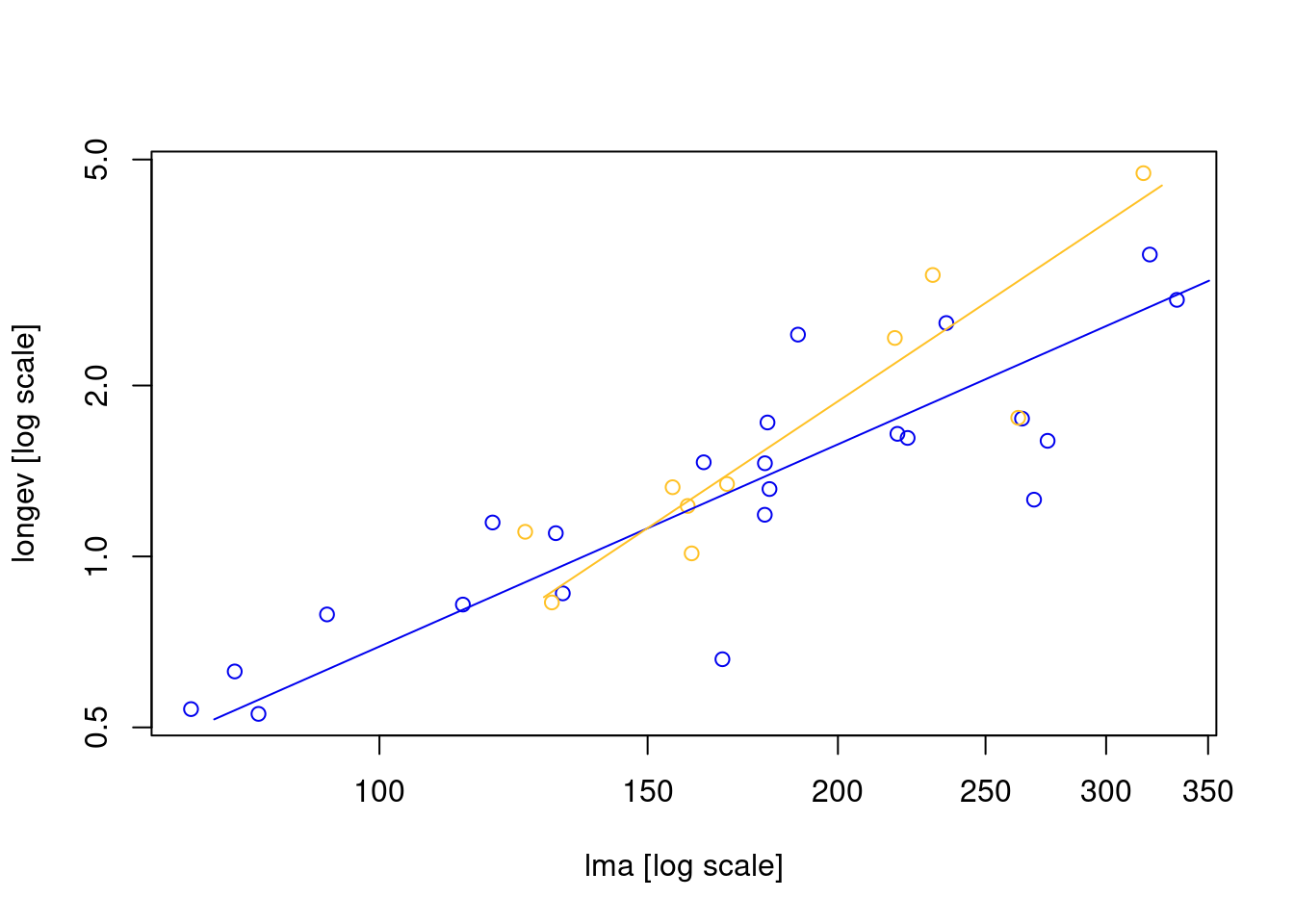
我们就无需在检验截距和延主轴的漂移了,差异足够明显。
26.2.3 多个样品无共同斜率
下面的数据时无共同斜率的情况,我就不一一解释了:
Code
Call: sma(formula = longev ~ lma * rain, data = low_soilp, log = "xy")
Fit using Standardized Major Axis
These variables were log-transformed before fitting: xy
Confidence intervals (CI) are at 95%
------------------------------------------------------------
Results of comparing lines among groups.
H0 : slopes are equal.
Likelihood ratio statistic : 2.367 with 1 degrees of freedom
P-value : 0.12395
------------------------------------------------------------
Coefficients by group in variable "rain"
Group: high
elevation slope
estimate -2.321737 1.1768878
lower limit -3.475559 0.7631512
upper limit -1.167915 1.8149286
H0 : variables uncorrelated.
R-squared : 0.3407371
P-value : 0.013891
Group: low
elevation slope
estimate -3.837710 1.786551
lower limit -5.291926 1.257257
upper limit -2.383495 2.538672
H0 : variables uncorrelated.
R-squared : 0.80651
P-value : 0.00041709 Code
plot(com_test)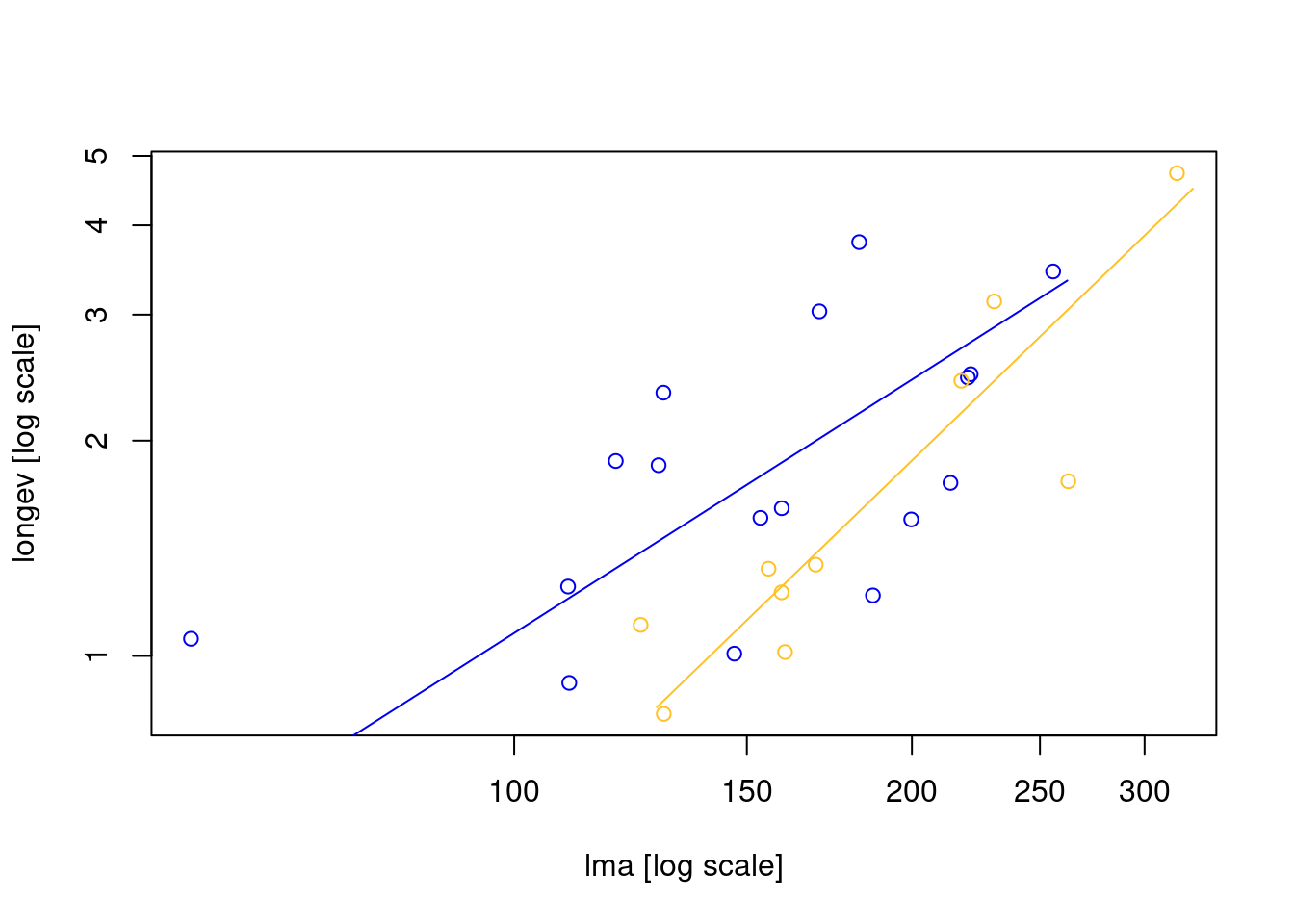
结论: H0 : slopes are equal. P 为 0.13,也就是接受 h0
Code
Call: sma(formula = longev ~ lma * rain, data = low_soilp, log = "xy",
slope.test = 1)
Fit using Standardized Major Axis
These variables were log-transformed before fitting: xy
Confidence intervals (CI) are at 95%
------------------------------------------------------------
Results of comparing lines among groups.
H0 : slopes are equal.
Likelihood ratio statistic : 2.367 with 1 degrees of freedom
P-value : 0.12395
------------------------------------------------------------
H0 : common slope not different from 1
Likelihood ratio statistic = 8.677 with 2 degrees of freedom under H0
P-value : 0.013057
Coefficients by group in variable "rain"
Group: high
elevation slope
estimate -2.321737 1.1768878
lower limit -3.475559 0.7631512
upper limit -1.167915 1.8149286
H0 : variables uncorrelated.
R-squared : 0.3407371
P-value : 0.013891
H0 : slope not different from 1
Test statistic: r= 0.1975 with 15 degrees of freedom under H0
P-value : 0.44733
Group: low
elevation slope
estimate -3.837710 1.786551
lower limit -5.291926 1.257257
upper limit -2.383495 2.538672
H0 : variables uncorrelated.
R-squared : 0.80651
P-value : 0.00041709
H0 : slope not different from 1
Test statistic: r= 0.8126 with 8 degrees of freedom under H0
P-value : 0.0042704 Code
Call: sma(formula = longev ~ lma + rain, data = low_soilp, log = "xy",
type = "elevation")
Fit using Standardized Major Axis
These variables were log-transformed before fitting: xy
Confidence intervals (CI) are at 95%
------------------------------------------------------------
Results of comparing lines among groups.
H0 : slopes are equal.
Likelihood ratio statistic : 2.367 with 1 degrees of freedom
P-value : 0.12395
------------------------------------------------------------
H0 : no difference in elevation.
Wald statistic: 6.566 with 1 degrees of freedom
P-value : 0.010393
------------------------------------------------------------
Coefficients by group in variable "rain"
Group: high
elevation slope
estimate -3.140896 1.551400
lower limit -4.079825 1.109374
upper limit -2.201966 2.011726
H0 : variables uncorrelated.
R-squared : 0.3407371
P-value : 0.013891
Group: low
elevation slope
estimate -3.304865 1.551400
lower limit -4.353328 1.109374
upper limit -2.256403 2.011726
H0 : variables uncorrelated.
R-squared : 0.80651
P-value : 0.00041709 Code
plot(low_soilp_elev)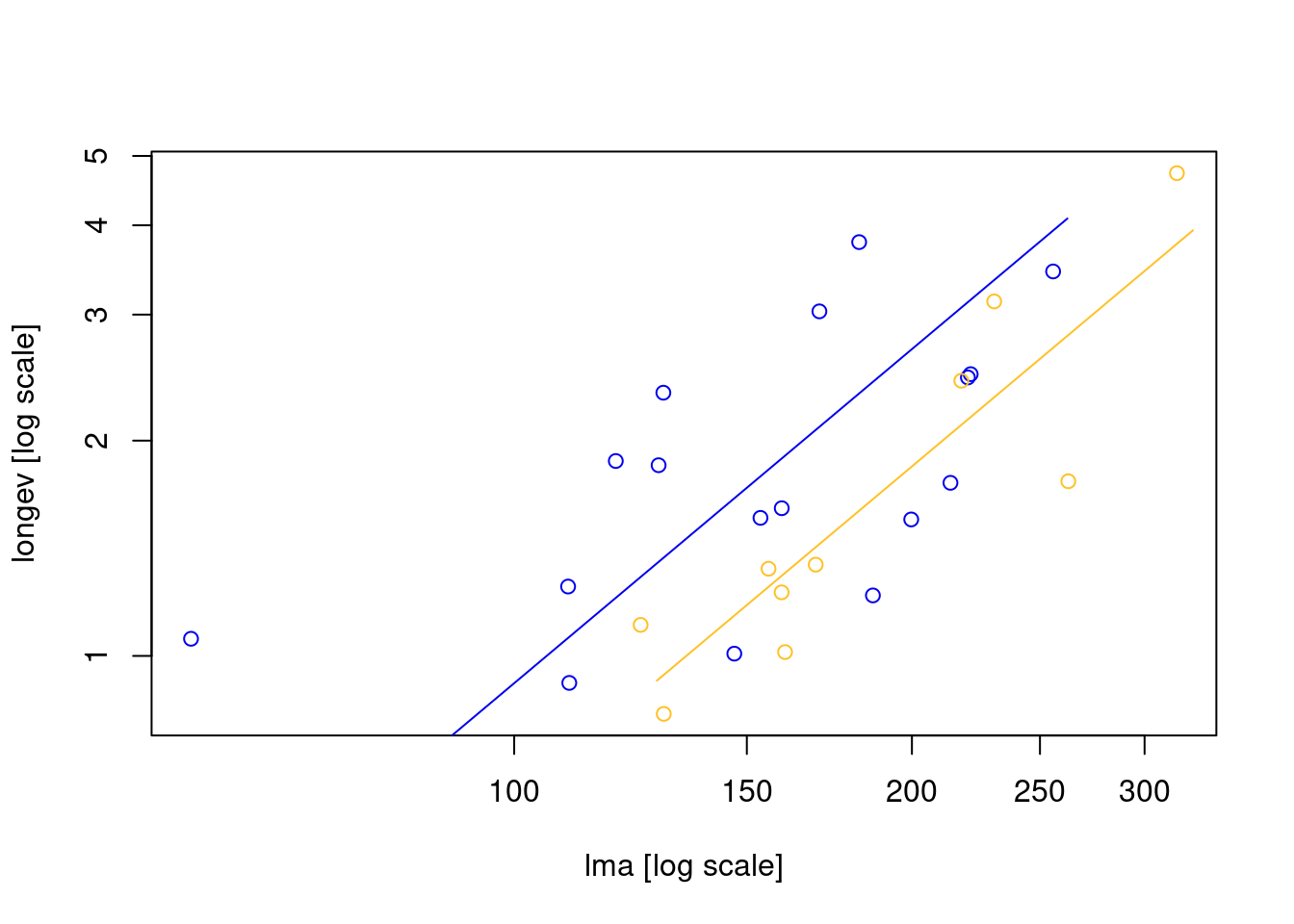
结论: H0 : no difference in elevation. p 为 0.01 共同截距有显著差异
Code
Call: sma(formula = longev ~ lma + rain, data = low_soilp, log = "xy",
type = "shift")
Fit using Standardized Major Axis
These variables were log-transformed before fitting: xy
Confidence intervals (CI) are at 95%
------------------------------------------------------------
Results of comparing lines among groups.
H0 : slopes are equal.
Likelihood ratio statistic : 2.367 with 1 degrees of freedom
P-value : 0.12395
------------------------------------------------------------
H0 : no shift along common axis.
Wald statistic: 0.2091 with 1 degrees of freedom
P-value : 0.64745
------------------------------------------------------------
Coefficients by group in variable "rain"
Group: high
elevation slope
estimate -3.140896 1.551400
lower limit -3.475559 1.109374
upper limit -1.167915 2.011726
H0 : variables uncorrelated.
R-squared : 0.3407371
P-value : 0.013891
Group: low
elevation slope
estimate -3.304865 1.551400
lower limit -5.291926 1.109374
upper limit -2.383495 2.011726
H0 : variables uncorrelated.
R-squared : 0.80651
P-value : 0.00041709 结论: H0 : no shift along common axis. p 为 0.64,接受 h0,无延主轴方向的漂移。