Code
remotes::install_github("zhujiedong/readphoto")
# 或
remotes::install_git("https://gitee.com/zhu_jie_dong/readphoto")下面的 R 软件包不再维护,建议参考@sec-6800-import-python中python图形界面的批量处理方式。
鉴于无论是 LI-6400 还是 LI-6800 数据 Excel 格式的复杂性,目前已知的能处理的软件包有 R 的 XLConnect 和 xlwings,文中对直接下载的 Excel 格式的数据转换,均依赖于此进行。
暂时只有我的 github repo 中的版本:
remotes::install_github("zhujiedong/readphoto")
# 或
remotes::install_git("https://gitee.com/zhu_jie_dong/readphoto")将所有 LI-6400 的 原始数据文件 放置于一个文件夹下,例如下面代码放在了名为 6400 的文件夹内,基本参数如下:
#| label: read_bat_6400
library(readphoto)
df64 <- read_bat_6400("data/6400", header_line = 17, data_start = 27)其中 header_line 是表头所在行,即 obs 开始的那一行的行号,data_start 也就是我们实际测量数据开始的行号,数据输出如下所示(仅显示前8列数据):
#| label: 6400kable
#| echo: false
kable(head(df64), caption = 'LI-6400 批量整合数据{#tbl-6400-importkable}')如果想另存为 csv 格式:
write.csv(df64, "./combined.csv")header_line 表示你数据表头所在行,data_start 表示你数据起始行,Obs = 1 时所在行,不含表头。这个也要确认好了,不同的测量不能放在一起(务必不要混淆,不然所有数据会放在一个文件夹内),不同的测量可能起始行不同,这样就会报错。
其中,数据的来源在表格中第一列,叫做 files,是数据来源的文件名(即你起的名字),例如本例中你看到的 aci 是我之前数据里面 aci 响应曲线的数据。
这些数据可以用于后文相关的分析中,尤其是像 fitacis 这样的函数,因为本质上他们都是符合 tidyverse 样式的数据。
参数的重计算函数为 recomp_6400, 其参数除了 read_bat_6400 所包含的参数外,还有叶面积 S, 以及叶片正反面的气孔比例,默认值分别为 6 和 0.5。
#| label: revise-6400-area
library(readphoto)
x1 <- read_bat_6400("./data/6400")
y1 <- recomp_6400("./data/6400", header_line = 17, data_start = 27, S = 6, K = 0.5)
x1$Photo - y1$Photo
x1$Trmmol - y1$Trmmol
x1$Cond - y1$Cond
x1$Ci-y1$Ci
# half of original the area
y1 <- recomp_6400("./data/6400", header_line = 17, data_start = 27, S = 3, K = 0.5)
y1$Photo/x1$Photo
# test with random area less than six
area <- 6 - runif(32, 1, 3)
y1 <- recomp_6400("./data/6400", header_line = 17, data_start = 27, S = area, K = 0.5)
y1$Photo/x1$Photo我们看到各个值之差非常小,因为我们使用的是相同的叶面积,理论上这两次读数的差异应为 0, 但在实际计算过程中,有小数点位数的影响,所以某些值不完全为 0,但该差值足够小。我们将所有的数据叶面积减半后,二者比值也约等于 2.
LI-6800 的数据我们可以直接处理 Excel 即可,我们分开来看两种方式:
XLConnect 直接读取功能比较麻烦,为了节省时间,我将其写成了函数,放在了我的 readphoto 软件包里,软件包的安装参考 sec. 5.2.2 :
当然,最近连我自己安装 github 的软件包都经常出问题,如果大家同样遇到问题,可以按照下面的方式安装:
它的参数很少,主要作用为:
path 是 Excel 文件的路径;
start_row 是数据开始的行号;
S 为修改的叶面积,默认值为 6,如果叶面积无需更改,则使用默认的 NULL。如果叶面积非默认值,该值必须输入一个长度和测量值数量完全一致的向量,例如,一共有三个测量值,只有第一个叶片没充满叶室,面积为 1.5,其他的为 2,则输入方式为 S = c(1.5, 2, 2)。
我们直接使用下面的例子解释,导入的数据是 6 cm2 的默认面积:
#| label: revise-6800-area
library(readphoto)
df6 <- xlconnect_read("./data/aci-xlc.xlsx")
df6$A
df3 <- xlconnect_read("./data/aci-xlc.xlsx", S = rep(3, 12))
df6$A/df3$A
df_random <- xlconnect_read("./data/aci-xlc.xlsx", S = rnorm(12, 3, 0.1))
df6$A/df_random$A光合速率的倍数的变化在预期之内。
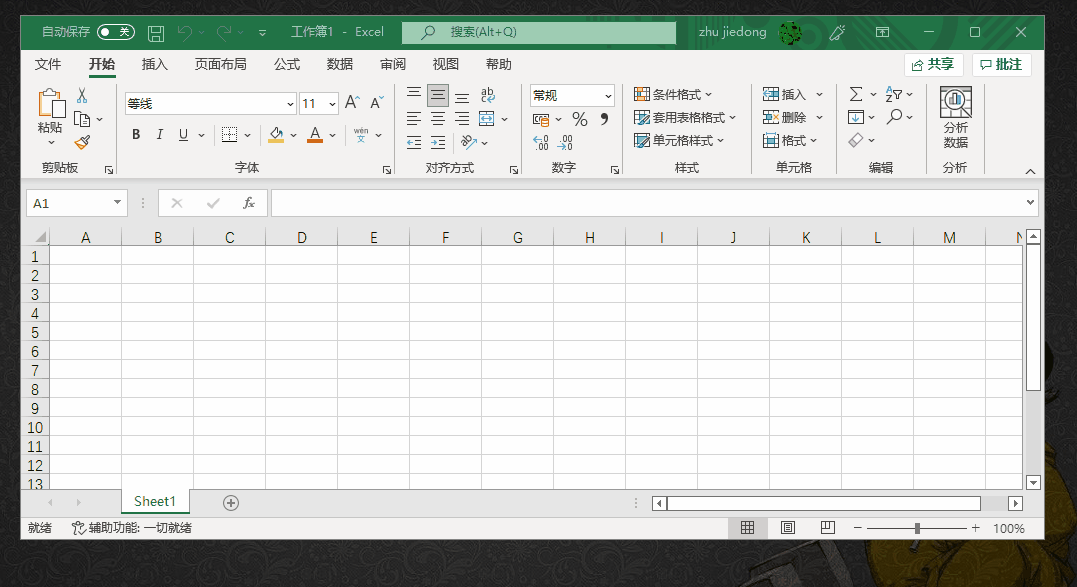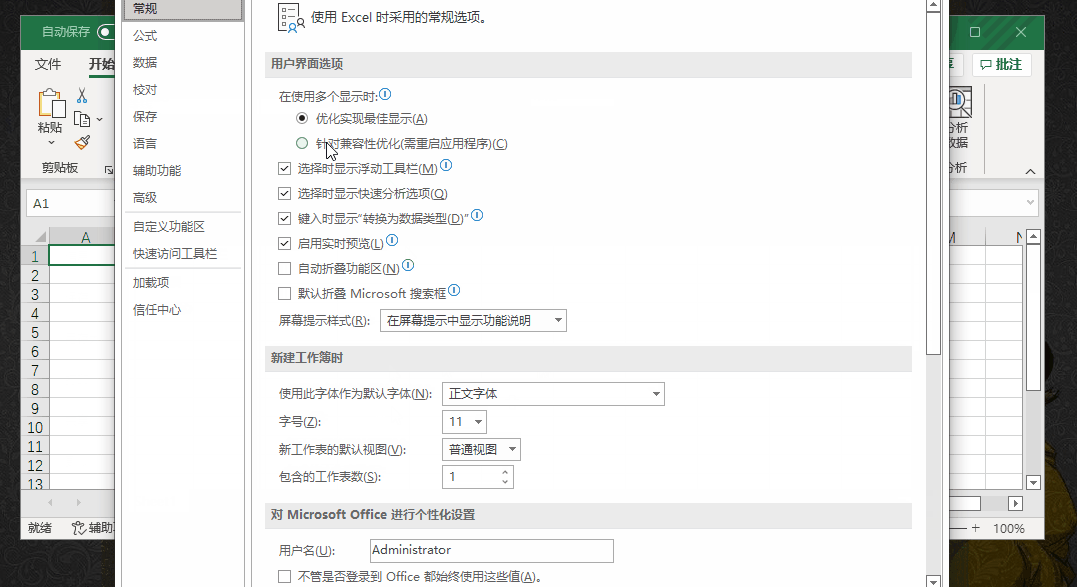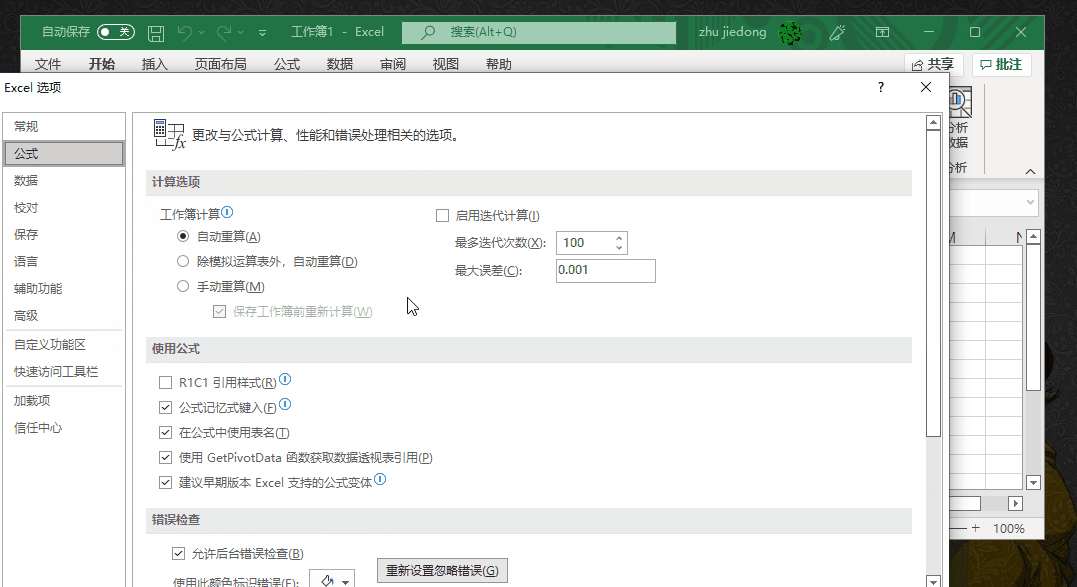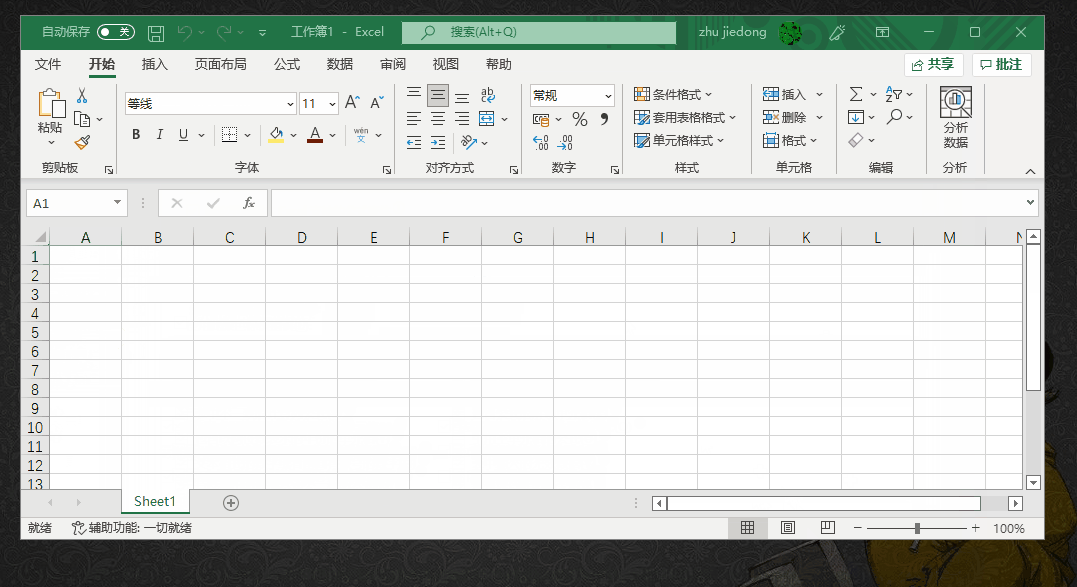
该功能需要启用 excel 的迭代计算,具体为文件 –> 选项–> 公式 –> 启动迭代计算,否则导入的带公式的单元格数值为 0。
# install.packages("devtools")
#devtools::install_github("zhujiedong/ReadLICOR6800")
remotes::install_git("https://gitee.com/zhu_jie_dong/ReadLICOR6800")使用比较简单,第一个函数用于读取单个数据文件:
library(ReadLICOR6800)
aci <- read_li6800('data/batch6800/racirtest1.xlsx')
aci[1:3, 1:6]
names(aci)
aci$data_tag默认的参数会添加 data_tag 列,用于读取多个文件时候区分数据用,默认的不坐修改的标签是文件名,若要修改,指定 tags 参数为我们需要的标签即可。
若单纯读取单个文件,也没有必要加标签,但加或者不加对我们分析数据没有任何影响:
与上面的函数类似,本身就是对其进行了包裹,方便不熟悉使用的人能够一次批量读取所有文件,需要注意所有文件的列数必须相同,这多数时候不会出现问题,多台光合仪同时使用时配置文件一致即可。否则只能分开读取再合并。使用 tags=NULL(默认), data_tag会显示文件名:
list.files("data/batch6800/")
aci <- read_li6800_folder("data/batch6800/")
names(aci)
head(aci$data_tag)
tail(aci$data_tag)当指定自定义标签时,需要 tags 给出的标签的长度和顺序要与 list.files() 显示的一致才可以。
aci <- read_li6800_folder("data/batch6800/", tags = paste('test', 1:2))
aci[1:3, 1:6]
names(aci)
head(aci$data_tag)
tail(aci$data_tag)也十分方便。
目前版本的使用非常简单,可参考:
下载连接:
# 百度网盘
链接:https://pan.baidu.com/s/18vi3jAd_T1m8UjAHa4TfoQ
提取码:1111
# 夸克网盘
链接:https://pan.quark.cn/s/3aa949607afd
# 阿里云盘
https://www.aliyundrive.com/s/43nmbKCqEve
如果还是比习惯用 R,我们来处理上面的 csv 文件即可 (期望后面添加到软件包里,成为一个函数,rSys.Date()` 尚未有时间修改):
files_csv <- list.files("./data/csvdata", full.names = TRUE)
files_csv[1] "./data/csvdata/racirtest1.csv" "./data/csvdata/rcirtest3.csv" 会在数据的添加 remarks,在最后一列,按照上面的代码,产生的内容是 csv 文件的文件名:
| obs | time | elapsed | date | hhmmss | averaging | TIME | E | Emm | A | Ca | Ci | Pci | Pca | gsw | gbw | gtw | gtc | Rabs | TleafEB | TleafCnd | SVPleaf | RHcham | VPcham | SVPcham | VPDleaf | LatHFlux | SenHFlux | NetTherm | EBSum | Asty | Esty | Adyn | Crd | Csd | dCsd.dt | αVc | Edyn | Hr | Hs | dHs.dt | αVh | Leak | LeakPct | CorrFact | CorrFactPct | Fan | DarkAdaptedID | Qmax_d | Fo | Fm | Fv.Fm | A_dark | LightAdaptedID | Qmax | Fs | Fm. | PhiPS2 | PS2.1 | Qabs_fs | A_fs | ETR | PhiCO2 | NPQ | alt..Fo. | DarkPulseID | Fmin | Fo. | Fv..Fm. | qP | qN | qP_Fo | qN_Fo | qL | X1.qL | Qin | Qabs | alpha | convert | S | K | Geometry | Custom | UseDynamic | TIME.1 | CO2_s | CO2_r | H2O_s | H2O_r | CO2_a | H2O_a | Flow | Pa | ΔPcham | Tair | Tleaf | Tleaf2 | Offset | Offset2 | Fan_speed | Qamb_in | Qamb_out | Q | f_red | f_blue | f_farred | F | Q_modavg | F_dc | Pc | Tled | TDigital | TPreamp | TPwrSpy | TDrive | Q_red | Q_blue | Q_farred | TSPF | state | F_avg | dF.dt | dF_dc.dt | F_dc_avg | period | time.1 | hhmmss.1 | co2_t | h2o_t | count | co2_adj | h2o_adj | co2_match | h2o_match | co2_at | h2o_at | co2_cv | h2o_cv | MatchValveR | MatchValveS | MatchCO2 | MatchH2O | cf_co2_a | cf_co2_b | cf_co2_c | cf_co2_d | cf_h2o_a | cf_h2o_b | cf_h2o_c | cf_h2o_d | co2_fit_low | co2_fit_high | h2o_fit_low | h2o_fit_high | co2_elapsed | h2o_elapsed | DIAG | Flow_s | Flow_r | Txchg | Tirga | Tchopper | Ts | Tr | CO2_. | Desiccant_. | Humidifier_. | Txchg_sp | CO2_r_sp | H2O_r_sp | SS_s | SS_r | remarks |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 1617082892 | 0.0 | 20210330 13:41:32 | 13:41:32 | 2 | 1617082891 | 0.0011770 | 1.177006 | -1.296405 | 21.91885 | 43.36186 | 4.386171 | 2.217152 | 0.0953076 | 2.949502 | 0.0936291 | 0.0586666 | 193.8115 | 27.66056 | 26.28095 | 3.430763 | 61.93396 | 2.194537 | 3.543350 | 1.236227 | -51.90596 | 87.13149 | 6.346983 | 235.3840 | -1.3088615 | 1.178213 | -1.296405 | 20.41723 | 22.40166 | -0.0000519 | 66.50013 | 1.177006 | 19.96748 | 21.69496 | -5.55e-05 | 79.88 | 0 | 0 | 1 | 0 | 53510.68 | - | - | 0 | 0 | NA | 0 | - | - | 0 | 0 | NA | 0.5 | 1009.183 | -1.296405 | NA | -0.0012846 | NA | NA | - | 0 | NA | NA | NA | NA | NA | NA | NA | NA | 1199.995 | 1009.183 | 0.8409898 | 0.1615102 | 9 | 0.5 | 0: Broadleaf | 2 | 1 | 1617082891 | 21.91885 | 19.99432 | 21.69527 | 19.96633 | 17.59828 | 21.71718 | 600.0092 | 101.1055 | 0.0472456 | 26.82890 | 26.28095 | 999.9 | 0 | 0 | 10003.595 | 0 | 45.29030 | 1199.995 | 0.967009 | 0.0329912 | 0 | 2.750250 | 0 | 4008.905 | 10372.25 | 40.73425 | 43.56250 | 42.42150 | 41.87475 | 40.79650 | 1160.405 | 39.59 | 0 | 1617082893 | 0 | 2.565665 | 0.4810222 | 17.091624 | 4006.983 | 15 | 1617082512 | 13:35:12 | 1617082511 | 1617082512 | 2 | -0.012 | -0.035 | 4.321 | -0.022 | 400 | 20 | 0.13 | 0.05 | 100 | 100 | 4.320 | -0.0219 | 4.32055 | 0 | 0 | 0 | -0.02194 | 0 | 0 | 0 | -1 | -1 | -1 | -1 | 6.4 | 6.3 | 18 | 646.822 | 392.817 | 24.9992 | 27.8989 | 30.0000 | 27.9067 | 27.9480 | 3.98628 | 23.5803 | 47.208 | 25 | 23.3772 | 20 | 96.8399 | 99.4131 | racirtest1 |
| 2 | 1617082895 | 2.5 | 20210330 13:41:35 | 13:41:35 | 2 | 1617082894 | 0.0011787 | 1.178677 | -1.313044 | 21.90930 | 43.60568 | 4.410859 | 2.216198 | 0.0954288 | 2.947543 | 0.0937450 | 0.0587395 | 193.8123 | 27.66178 | 26.28185 | 3.430946 | 61.92849 | 2.194488 | 3.543584 | 1.236458 | -51.97964 | 87.10935 | 6.349654 | 235.2916 | -1.3202228 | 1.179134 | -1.313044 | 20.38174 | 22.39189 | -0.0001096 | 66.50013 | 1.178677 | 19.96514 | 21.69502 | -4.55e-05 | 79.88 | 0 | 0 | 1 | 0 | 53453.39 | - | - | 0 | 0 | NA | 0 | - | - | 0 | 0 | NA | 0.5 | 1009.188 | -1.313044 | NA | -0.0013011 | NA | NA | - | 0 | NA | NA | NA | NA | NA | NA | NA | NA | 1200.000 | 1009.188 | 0.8409898 | 0.1615102 | 9 | 0.5 | 0: Broadleaf | 2 | 1 | 1617082894 | 21.90930 | 19.96775 | 21.69468 | 19.96438 | 17.58877 | 21.71660 | 600.0103 | 101.1068 | 0.0465612 | 26.83002 | 26.28185 | 999.9 | 0 | 0 | 9992.340 | 0 | 45.41602 | 1200.000 | 0.967009 | 0.0329912 | 0 | 2.646775 | 0 | 4009.425 | 10372.30 | 40.75000 | 43.53100 | 42.40600 | 41.87450 | 40.81200 | 1160.410 | 39.59 | 0 | 1617082895 | 0 | 2.575250 | 0.3805983 | 16.909402 | 4007.662 | 15 | 1617082512 | 13:35:12 | 1617082511 | 1617082512 | 2 | -0.012 | -0.035 | 4.321 | -0.022 | 400 | 20 | 0.13 | 0.05 | 100 | 100 | 4.320 | -0.0219 | 4.32055 | 0 | 0 | 0 | -0.02194 | 0 | 0 | 0 | -1 | -1 | -1 | -1 | 6.4 | 6.4 | 18 | 647.002 | 392.759 | 24.9990 | 27.8989 | 30.0000 | 27.9056 | 27.9480 | 4.08099 | 23.5803 | 47.208 | 25 | 26.7963 | 20 | 96.8405 | 99.4111 | racirtest1 |
| 3 | 1617082896 | 4.0 | 20210330 13:41:36 | 13:41:36 | 2 | 1617082895 | 0.0011796 | 1.179583 | -1.321542 | 21.91320 | 43.73144 | 4.423538 | 2.216572 | 0.0955220 | 2.947691 | 0.0938351 | 0.0587960 | 193.8119 | 27.66123 | 26.28070 | 3.430713 | 61.92991 | 2.194503 | 3.543527 | 1.236210 | -52.01960 | 87.25278 | 6.359744 | 235.4048 | -1.2793724 | 1.179630 | -1.321542 | 20.38576 | 22.40783 | 0.0001313 | 66.50013 | 1.179583 | 19.96436 | 21.69522 | 1.69e-05 | 79.88 | 0 | 0 | 1 | 0 | 53457.74 | - | - | 0 | 0 | NA | 0 | - | - | 0 | 0 | NA | 0.5 | 1009.186 | -1.321542 | NA | -0.0013095 | NA | NA | - | 0 | NA | NA | NA | NA | NA | NA | NA | NA | 1199.997 | 1009.186 | 0.8409898 | 0.1615102 | 9 | 0.5 | 0: Broadleaf | 2 | 1 | 1617082895 | 21.91320 | 20.03295 | 21.69503 | 19.96400 | 17.59267 | 21.71698 | 600.0110 | 101.1058 | 0.0466098 | 26.82975 | 26.28070 | 999.9 | 0 | 0 | 9993.280 | 0 | 45.47442 | 1199.997 | 0.967009 | 0.0329912 | 0 | 2.729575 | 0 | 4009.827 | 10372.27 | 40.73425 | 43.56225 | 42.39050 | 41.87475 | 40.79650 | 1160.408 | 39.59 | 0 | 1617082897 | 0 | 2.576960 | 0.7380154 | 16.976923 | 4008.236 | 15 | 1617082512 | 13:35:12 | 1617082511 | 1617082512 | 2 | -0.012 | -0.035 | 4.321 | -0.022 | 400 | 20 | 0.13 | 0.05 | 100 | 100 | 4.321 | -0.0219 | 4.32055 | 0 | 0 | 0 | -0.02194 | 0 | 0 | 0 | -1 | -1 | -1 | -1 | 6.4 | 6.4 | 18 | 646.866 | 392.795 | 24.9990 | 27.8989 | 30.0000 | 27.9056 | 27.9471 | 4.17522 | 23.5803 | 47.208 | 25 | 26.7963 | 20 | 96.8409 | 99.4112 | racirtest1 |
| 4 | 1617082899 | 6.5 | 20210330 13:41:39 | 13:41:39 | 2 | 1617082898 | 0.0011813 | 1.181340 | -1.280420 | 21.99530 | 43.07574 | 4.357142 | 2.224840 | 0.0957200 | 2.948978 | 0.0940269 | 0.0589164 | 193.8135 | 27.65659 | 26.27753 | 3.430070 | 61.94557 | 2.194561 | 3.542725 | 1.235509 | -52.09708 | 87.18356 | 6.351701 | 235.2516 | -0.7424130 | 1.181656 | -1.280420 | 20.90404 | 22.55299 | 0.0691623 | 66.50013 | 1.181340 | 19.96294 | 21.69646 | -2.70e-06 | 79.88 | 0 | 0 | 1 | 0 | 53495.89 | - | - | 0 | 0 | NA | 0 | - | - | 0 | 0 | NA | 0.5 | 1009.194 | -1.280420 | NA | -0.0012688 | NA | NA | - | 0 | NA | NA | NA | NA | NA | NA | NA | NA | 1200.007 | 1009.194 | 0.8409897 | 0.1615102 | 9 | 0.5 | 0: Broadleaf | 2 | 1 | 1617082898 | 21.99530 | 20.92070 | 21.69595 | 19.96197 | 17.67475 | 21.71790 | 600.0185 | 101.1040 | 0.0467119 | 26.82590 | 26.27753 | 999.9 | 0 | 0 | 10000.763 | 0 | 45.54863 | 1200.007 | 0.967009 | 0.0329912 | 0 | 2.628825 | 0 | 4009.780 | 10372.38 | 40.74950 | 43.59350 | 42.40600 | 41.96850 | 40.81200 | 1160.418 | 39.59 | 0 | 1617082900 | 0 | 2.605660 | 0.9306615 | 13.372308 | 4008.739 | 15 | 1617082512 | 13:35:12 | 1617082511 | 1617082512 | 2 | -0.012 | -0.035 | 4.321 | -0.022 | 400 | 20 | 0.13 | 0.05 | 100 | 100 | 4.320 | -0.0219 | 4.32055 | 0 | 0 | 0 | -0.02194 | 0 | 0 | 0 | -1 | -1 | -1 | -1 | 6.5 | 6.5 | 18 | 646.672 | 392.887 | 24.9989 | 27.8981 | 30.0000 | 27.9056 | 27.9457 | 4.38255 | 23.5803 | 47.208 | 25 | 33.5145 | 20 | 96.8411 | 99.4125 | racirtest1 |
| 5 | 1617082900 | 8.0 | 20210330 13:41:40 | 13:41:40 | 2 | 1617082899 | 0.0011824 | 1.182408 | -1.283279 | 22.19683 | 43.29043 | 4.378882 | 2.245237 | 0.0958617 | 2.947640 | 0.0941628 | 0.0590019 | 193.8127 | 27.65527 | 26.27460 | 3.429477 | 61.95251 | 2.194626 | 3.542433 | 1.234851 | -52.14418 | 87.38634 | 6.369227 | 235.4241 | -0.1925011 | 1.182614 | -1.283279 | 21.85036 | 22.91441 | 0.2003223 | 66.50013 | 1.182408 | 19.96182 | 21.69676 | 3.49e-05 | 79.88 | 0 | 0 | 1 | 0 | 53457.15 | - | - | 0 | 0 | NA | 0 | - | - | 0 | 0 | NA | 0.5 | 1009.190 | -1.283279 | NA | -0.0012716 | NA | NA | - | 0 | NA | NA | NA | NA | NA | NA | NA | NA | 1200.003 | 1009.190 | 0.8409897 | 0.1615102 | 9 | 0.5 | 0: Broadleaf | 2 | 1 | 1617082899 | 22.19683 | 21.94745 | 21.69647 | 19.96105 | 17.87627 | 21.71840 | 600.0030 | 101.1045 | 0.0467717 | 26.82450 | 26.27460 | 999.9 | 0 | 0 | 9993.112 | 0 | 45.56580 | 1200.003 | 0.967009 | 0.0329912 | 0 | 2.624775 | 0 | 4009.068 | 10372.33 | 40.71850 | 43.64025 | 42.40600 | 41.98400 | 40.79650 | 1160.412 | 39.59 | 0 | 1617082901 | 0 | 2.623516 | 0.1101154 | 9.382308 | 4008.880 | 15 | 1617082512 | 13:35:12 | 1617082511 | 1617082512 | 2 | -0.012 | -0.035 | 4.321 | -0.022 | 400 | 20 | 0.13 | 0.05 | 100 | 100 | 4.320 | -0.0219 | 4.32055 | 0 | 0 | 0 | -0.02194 | 0 | 0 | 0 | -1 | -1 | -1 | -1 | 6.5 | 6.5 | 18 | 646.846 | 392.828 | 24.9988 | 27.8972 | 29.9999 | 27.9056 | 27.9457 | 4.46969 | 23.5803 | 47.208 | 25 | 33.5145 | 20 | 96.8413 | 99.4125 | racirtest1 |
| 6 | 1617082902 | 10.0 | 20210330 13:41:42 | 13:41:42 | 2 | 1617082902 | 0.0011830 | 1.183029 | -1.302558 | 23.10023 | 44.45623 | 4.496921 | 2.336679 | 0.0960590 | 2.947577 | 0.0943531 | 0.0591215 | 193.8133 | 27.65226 | 26.26610 | 3.427756 | 61.96520 | 2.194706 | 3.541836 | 1.233051 | -52.17157 | 88.27968 | 6.434109 | 236.3555 | 1.1688374 | 1.183105 | -1.302558 | 23.99017 | 23.88864 | 0.4662872 | 66.50013 | 1.183029 | 19.96064 | 21.69664 | 1.80e-05 | 79.88 | 0 | 0 | 1 | 0 | 53455.89 | - | - | 0 | 0 | NA | 0 | - | - | 0 | 0 | NA | 0.5 | 1009.193 | -1.302558 | NA | -0.0012907 | NA | NA | - | 0 | NA | NA | NA | NA | NA | NA | NA | NA | 1200.007 | 1009.193 | 0.8409897 | 0.1615102 | 9 | 0.5 | 0: Broadleaf | 2 | 1 | 1617082902 | 23.10023 | 24.89453 | 21.69670 | 19.96050 | 18.77970 | 21.71863 | 599.9837 | 101.1070 | 0.0468953 | 26.82163 | 26.26610 | 999.9 | 0 | 0 | 9992.510 | 0 | 45.60887 | 1200.007 | 0.967009 | 0.0329912 | 0 | 2.594067 | 0 | 4006.967 | 10372.37 | 40.75000 | 43.54167 | 42.39567 | 41.79133 | 40.79133 | 1160.417 | 39.59 | 0 | 1617082903 | 0 | 2.620524 | 0.0663154 | -4.953846 | 4008.747 | 15 | 1617082512 | 13:35:12 | 1617082511 | 1617082512 | 2 | -0.012 | -0.035 | 4.321 | -0.022 | 400 | 20 | 0.13 | 0.05 | 100 | 100 | 4.320 | -0.0219 | 4.32055 | 0 | 0 | 0 | -0.02194 | 0 | 0 | 0 | -1 | -1 | -1 | -1 | 6.5 | 6.5 | 18 | 646.938 | 392.756 | 24.9988 | 27.8965 | 30.0000 | 27.9051 | 27.9457 | 4.62190 | 23.5803 | 47.208 | 25 | 36.8578 | 20 | 96.8411 | 99.4118 | racirtest1 |
到此为止已经整理为所谓的 tidy data 了,用 tidyverse 也好,用 base 语法也好,总之是比较容易处理的数据了,例如上面其实是 RACiR 数据了,有两个,那么当然可以使用 plantecophys::fitacis 来一条命令搞定所有数据的拟合了。
这里需要注意的是使用 xlconnect_read 也可以使用类似的操作,但我觉得不如这种方法省事,故而只列出来这一种,有需要的也可以按照类似方法处理即可。